Описание
Сайт узнавания и позиция гидролиза:
| R(5mC)GY | R(5mC)↑GY |
| YG(5mC)R | YG↓(5mC)R |
Источник: Из штамма E.coli, несущего клонированный ген Gla I из Glacial ice bacterium Gl29
Определение единицы активности:
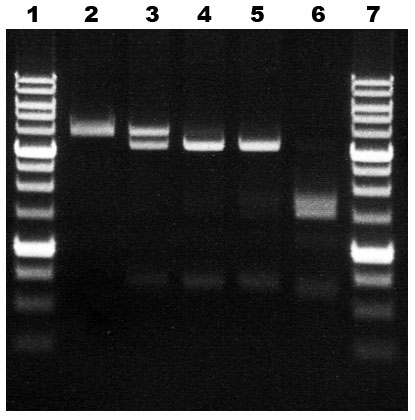
За единицу активности принимается количество фермента, необходимое для полного гидролиза 1 мкг плазмиды pHspAI2/GsaI по единственному сайту 5`-G(5mC)G(5mC)-3`/3`-(5mC)G(5mC)G-5` за 1 час при 30°С в 50 мкл реакционной смеси. При этом происходит исчезновение исходной линейной формы плазмиды pHspAI2/GsaI и образование двух основных (3,419 и 699 п.н.) фрагментов ДНК (см. рисунок ниже, дор. 3-5). Появление дополнительных продуктов гидролиза (см. рисунок ниже, дор. 6) является результатом последующего расщепления ДНК по сайтам 5`-GCGC-3`/3`-CGCG-5`, имеющим три и два 5-метилцитозина.
Определение активности GlaI
| В каждом случае проводилась инкубация фермента с 1 мкг ДНК pHspAI2/GsaI в 50 мкл реакционной смеси (см. Единица активности). После инкубации аликвота реакционной смеси 15 мкл наносилась на гель. Дорожки: 2 – Контрольная ДНК pHspAI2/GsaI, 3 – 0.5 мкл GlaI (разб. 1/100), 4 – 1 мкл GlaI (разб. 1/100), 5 – 2 мкл GlaI (разб. 1/100), 6 – 1 мкл неразбавленного фермента GlaI, 1 и 7- 1 Kb SE ДНК-маркеры. Продукты разделены в 1%-ном агарозном геле в буфере TAE. Продукты разделены в 1%-ном агарозном геле в буфере TAE. |
 В присутствии 20% DMSO активность фермента значительно возрастает по всем сайтам без потери специфичности В присутствии 20% DMSO активность фермента значительно возрастает по всем сайтам без потери специфичности |
Субстрат для определения активности:
Субстрат pHspAI2/GsaI представляет собой ДНК плазмиды pHspAI2, линеаризованной эндонуклеазой рестрикции GsaI. Плазмида pHspAI2 содержит фрагмент геномной ДНК бактериального штамма Haemophilus species A1, включающий ген ДНК-метилтрансферазы M.HspAI, и единственный канонический сайт узнавания GlaI 5`-G(5mC)G(5mC)-3`/3`-(5mC)G(5mC)G-5`[2].
Оптимальный буфер: SE-буфер Y
Активность фермента в различных буферах в процентах от максимальной:
| B | G | O | W | Y | ROSE |
| 75 | 75 | 25 | 25 | 100 | 100 |
Условия хранения: 10 mM Tris-HCl (pH 7.6); 250 mM NaCl; 0.1 mM EDTA; 7 mM 2-меркаптоэтанол; 0,1% Triton X-100, 50 мкг/мл BSA, 50% глицерин. Хранить при -20°С.
Неспецифический гидролиз: Не наблюдается неспецифического гидролиза при инкубации 100 ед фермента Gla I с 1 мкг ДНК фага лямбда в течение 16 часов при 30°С в 50 мкл реакционной смеси.
Чувствительность к метилированию:
Фермент расщепляет только С5-метилированную ДНК. Не расщепляет ДНК, содержащую N4-метилцитозин [1].
С ферментом поставляется: 10 Х SE-буфер Y, плазмида pHspAI2/GsaI.
Примечание:
Литература:
- Землянская Е. В., Дегтярев С. Х. Субстратная специфичность и свойства метилзависимых сайт-специфических ДНК-эндонуклеаз.// Молекулярная биология, том 47, № 6, с. 900–913(2013)
-
 Тарасова Г.В., Наякшина Т.Н., Дегтярев С.Х. Субстратная специфичность новой ДНК-эндонуклеазы GlaI // BMC Molecular Biology 2008, 9:7 – на английском языке
Тарасова Г.В., Наякшина Т.Н., Дегтярев С.Х. Субстратная специфичность новой ДНК-эндонуклеазы GlaI // BMC Molecular Biology 2008, 9:7 – на английском языке -
![]() Томилова Ю.Э., Чернухин В.А., Дегтярев С.Х. Зависимость активности сайт-специфической эндонуклеазы GlaI от количества и положения метилированных цитозинов в узнаваемой последовательности 5’-GCGC-3’. // Вестник биотехнологии и физико-химической биологии имени Ю.А.Овчинникова, 2006, т.2, №1, с. 30-39.
Томилова Ю.Э., Чернухин В.А., Дегтярев С.Х. Зависимость активности сайт-специфической эндонуклеазы GlaI от количества и положения метилированных цитозинов в узнаваемой последовательности 5’-GCGC-3’. // Вестник биотехнологии и физико-химической биологии имени Ю.А.Овчинникова, 2006, т.2, №1, с. 30-39.  Чернухин В.А. , Наякшина Т.Н., Абдурашитов М.А., Томилова Ю.Э., Мезенцева Н.В., Дедков В.С., Михненкова Н.А., Гончар Д.А., Дегтярев С.Х Новая эндонуклеаза рестрикции GlaI узнает метилированную последовательность 5’-G(5mC)^GC-3’. // Биотехнология, 2006, № 4, С.31-35
Чернухин В.А. , Наякшина Т.Н., Абдурашитов М.А., Томилова Ю.Э., Мезенцева Н.В., Дедков В.С., Михненкова Н.А., Гончар Д.А., Дегтярев С.Х Новая эндонуклеаза рестрикции GlaI узнает метилированную последовательность 5’-G(5mC)^GC-3’. // Биотехнология, 2006, № 4, С.31-35- Чернухин В.А., Наякшина Т.Н., Мезенцева Н.В., Томилова Ю.Э., Дегтярев С.Х., Дедков В.С. Штамм бактерий Glacial ice bacterium I – продуцент эндонуклеазы рестрикции Gla I.//Патент на изобретение RU 2287012 С1 (2006)
Применение:
- , , , , , , , , , , , , , , , , , , , , Early Diagnosis of Colorectal Cancer Based on Bisulfite-free Site-specific Methylation Identification PCR Strategy: High-Sensitivity, Accuracy, and Primary Medical Accessibility. Adv. Sci. 2024, 2401137. https://doi.org/10.1002/advs.202401137
- Qiaomin Wu, Yang Yu, Mengqi Chen, Jinyan Long, Xiaolan Yang A label-free fluorescence sensing strategy based on GlaI-assisted EXPAR for rapid and accurate quantification of human methyltranferase activity // Talanta, 269, 125456 (2024)
- Xu, G., Yang, H., Qiu, J. et al. Sequence terminus dependent PCR for site-specific mutation and modification detection.// Nature Communications 14, 1169 (2023)
- Zhou S, Sun H, Huo D, Wang X, Qi N, Peng L, Yang M, Lu P, Hou C.A novel methyl-dependent DNA endonuclease GlaI coupling with double cascaded strand displacement amplification and CRISPR/Cas12a for ultra-sensitive detection of DNA methylation.// Analytica Chimica Acta, Volume 1212, 15 June 2022
- Hao Yang, Jiani Qiu, LinQing Zhen, Yizhou Huang, Wei Ren, Hongchen Gu, Hong Xu, Gaolian Xu.Sensitive GlaI digestion and terminal transferase PCR for DNA methylation detection// Talanta 2022 Sep 1;247:123616 (2022)
- Dong N, Wang W, Qin Y, Wang Y, Shan H.Sensitive lateral flow assay for bisulfite-free DNA methylation detection based on the restriction endonuclease GlaI and rolling circle amplification// Analytica Chimica Acta, Volume 1227, 22 September 2022
- Wang LJ, Han X, Qiu JG, Jiang B, Zhang CY.Cytosine-5 methylation-directed construction of a Au nanoparticle-based nanosensor for simultaneous detection of multiple DNA methyltransferases at the single-molecule level// Chem Sci. 2020, 11, 9675-9684 (2020)
- Petrova, D. V., Naumenko, M. B., Khantakova, D. V., Grin, I. R. & Zharkov, D. O. Relative Efficiency of Recognition of 5-Methylcytosine and 5-Hydroxymethylcytosine by Methyl-Dependent DNA Endonuclease GlaI// Russian Journal of Bioorganic Chemistry T. 45, № 6, стр. 625-629. (2019)
- Susan M Mitchell, Keith N Rand, Zheng-Zhou Xu, Thu Ho, Glenn S Brown, Jason P Ross, Peter L Molloy Helper-Dependent Chain Reaction (HDCR) for Selective Amplification of Methylated DNA Sequences // Methods Mol Biol (2018)
- Yueying Sun, Yuanyuan Sun, Weimin Tian, Chenghui Liu, * Kejian Gao and Zhengping L A novel restriction endonuclease GlaI for rapid and highly sensitive detection of DNA methylation coupled with isothermal exponential amplification reaction // Chemical Science, 9, pp.1344-1351 (2018)
- Viktor Tomilov, Murat Abdurashitov, Danila Gonchar, Anastasiya Snezhkina, George Krasnov, Anna Kudryavtseva and Sergey Degtyarev Comparative analysis of RCGY sites methylation in the genomes of Raji and U-937 malignant and normal lung fibroblast cell lines // Cancer Epigenetics and Biomarkers, Osaka, Japan, October 2017
- A.G. Akishev et al. GLAD-PCR assay of selected R(5mC)GY sites in URB1 and CEPBD genes in human genome// Res J Pharm Biol Chem Sci, 8(1): pp.465-475, (2017)
- Evdokimov et al. GLAD-PCR Assay of DNA Methylation Markers Associated with Colorectal Cancer// Biol Med (Aligarh) , 8:7(2016)
- А.А.Евдокимов, Н.А.Нетесова, Н.А.Сметанникова, М.А.Абдурашитов, А.Г.Акишев, Е.С.Давидович, В.В. Кузнецов, Ю.Д.Ермолаев, А.Б.Карпов, А.Э.Сазонов, Р.М.Тахауов, С.Х.Дегтярев Применение метода GLAD-ПЦР анализа для выявления сайтов метилирования в регуляторных областях генов-онкосупрессоров ELMO1 и ESR1 при колоректальном раке // Вопросы онкологии, №1, стр. 116-120 (2016)
- EV Dubinin, AG Akishev, MA Abdurashitov, SB Oleynikova, VL Sitko, and S Kh Degtyarev Real time GlaI-PCR assay of regulation regions of human genes HDAC4, RARB and URB1 // Research Journal of Pharmaceutical, Biological and Chemical Sciences, vol 7(2), p.p. 667-676 (2016).
- Murat A . Abdurashitov , Valery A . Chernukhin , Danila A . Gonchar , Vladimir S . Dedkov, Natalia A . Mikhnenkova, and Sergey Kh . Degtyarev Dimethyl Sulfoxide Changes the Recognition Site Preference of Methyl- directed Site-specific DNA Endonuclease GlaI// Research Journal of Pharmaceutical, Biological and Chemical Sciences, № 7(1),с. 1733-1739 (2016)
- Abdurashitov M.A., Tomilov V.N., Gonchar D.A., Kuznetsov V.V., Degtyarev S.Kh. Mapping of R(5mC)GY Sites in the Genome of Human Malignant Cell Line Raji // Biol Med (Aligarh) Volume 7, Issue 4, BM-135-15 (2015)
- Кузнецов В.В., Абдурашитов М.А., Акишев А.Г., Дегтярев С.Х. Способ определения нуклеотидной последовательности Pu(5mC)GPy в заданном положении протяженной ДНК.// Патент на изобретение RU 2525710 С1 (2014)
- Fagan RL, Wu M, Chedin F, Brenner C An Ultrasensitive High Throughput Screen for DNA Methyltransferase 1-Targeted Molecular Probes //PLoS ONE 8(11): e78752. doi:10.1371/journal.pone.0078752 (2013)
- Keith N. Rand, Graeme P. Young, Thu Ho and Peter L. Molloy, Sensitive and selective amplification of methylated DNA sequences using helper-dependent chain reaction in combination with a methylationdependent restriction enzymes. // Nucleic Acids Research, pp. 1-10 (2012).
- F. Syeda, R.L. Fagan, M. Wean, G.V. Awakumov, J.R. Walker, S. Xue, S. Dhe-Paganon, & C. Brenner, The RFTS Domain is a DNA-competitive Inhibitor of Dnmt1//, JBC, v. 286, pp. 15344-15351 (2011).
- А.Г. Акишев, Д.А. Гончар, М.А. Абдурашитов, С.Х. Дегтярев Эпигенетическое типирование малигнантных клеточных линий человека с помощью BlsI- и GlaI ПЦР анализа // Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова. – 2011. – Т. 7 – № 3 – С. 5 – 16
- Kravets AP, Mousseau TA, Litvinchuk AV, Ostermiller Sh, Vengen GS, Grodzinski DM. Changes in wheat DNA methylation pattern after chronic seed gamma-irradiation.// Tsitol Genet. 2010 Sep-Oct;44(5):18-22. Russian.
- Wood, R. J., McKelvie, J. C., Maynard-Smith, M. D., and Roach, P. L. A real-time assay for CpG-specific cytosine-C5 methyltransferase activity.// (2010) Nucleic Acids Res 38, e107
- Д.А. Гончар, А.Г. Акишев, С.Х. Дегтярев BlsI- и GlaI- ПЦР анализ – новый метод исследования метилированных участков ДНК // Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова. – 2010. – Т. 6 – № 1 – С. 5-12
- Абдурашитов М.А., Чернухин В.А., Гончар Д.А., Дегтярев С.Х. GlaI digestion of mouse γ-satellite DNA: study of primary structure and ACGT sites methylation // BMC Genomics 2009, 10:322. – на английском языке
- В.А. Чернухин, М.А. Абдурашитов, В.Н. Томилов, Д.А. Гончар, С.Х. Дегтярев Сравнительный рестрикционный анализ хромосомной ДНК мыши in vitro и in silico // Вестник биотехнологии и физико-химической биологии имени Ю. А. Овчинникова, 2007, 3, №4, с. 19-27.



